Lección 6 Data frames
Habitualmente, dispondremos de una serie de datos que describirán algunos aspectos de un conjunto de individuos, y querremos analizarlos. El análisis estadístico de estos datos puede ser de dos tipos básicos:
Análisis exploratorio, o descriptivo, cuando nuestro objetivo sea resumir, representar y explicar los datos concretos de los que disponemos. La estadística descriptiva es el conjunto de técnicas que se usan con este fin.
Análisis inferencial, si nuestro objetivo es deducir (inferir), a partir de estos datos, información significativa sobre el total de la población o las poblaciones de interés. Las técnicas que se usan en este caso forman la estadística inferencial.
En las próximas lecciones explicaremos algunas técnicas básicas de estadística descriptiva orientadas al análisis de datos. Estas técnicas consistirán en una serie de medidas, gráficos y modelos descriptivos que nos permitirán resumir y explorar un conjunto de datos, con el objetivo final de entenderlos lo mejor posible. De todas formas, ambos tipos de análisis estadístico están relacionados. Así, por un lado, cualquier análisis inferencial se suele empezar explorando los datos que se usarán, y por otro, muchas técnicas descriptivas permiten estimar propiedades de la población de la que se ha extraído la muestra. Por citar un ejemplo, la media aritmética de las alturas de una muestra de individuos nos da un valor supuestamente representativo de esta muestra, pero también estima la media de las alturas del total de la población, si la muestra es aleatoria.
Los datos de los que disponemos suelen ser multidimensionales, en el sentido de que observamos varias características de una serie de individuos. Estos datos se tienen que registrar de alguna manera. Normalmente, los guardaremos en un archivo de ordenador con un formato preestablecido. Los formatos de almacenamiento de datos en un ordenador son diversos: texto simple (codificado en diferentes formatos: ASCII, isolatin, utf8…), hojas de cálculo (archivos de Open Office o Excel), bases de datos, etc. Una de las maneras básicas de almacenar datos es en forma de tablas de datos, pequeñas bases de datos donde se han anotado los valores de algunas variables para una serie de observaciones.
Como ya comentamos en la Lección 3, la manera más conveniente de guardar en R una tabla de datos es en forma de data frame. En concreto, un data frame es una tabla de doble entrada, formada por variables en las columnas y observaciones de estas variables en las filas, de manera que cada fila contiene los valores de las variables para un mismo caso o individuo. En este sentido, un data frame tiene la apariencia de una matriz, pero con la diferencia de que cada columna de un data frame puede contener datos de un tipo diferente, siempre que todos los datos de una misma columna sean del mismo tipo, porque corresponden a observaciones de una misma propiedad: así, una columna puede estar formada por números, por ejemplo, alguna medida; otra, por palabras, por ejemplo, la especie del individuo; otra, por valores lógicos, por ejemplo, que describan si una cierta propiedad está presente o ausente en el individuo; etc. De esta manera, las columnas de un data frame son vectores o factores, mientras que las filas son listas heterogéneas.
Los tipos de datos que consideramos en este curso son los siguientes:
Datos de tipo atributo, o cualitativos. Son los que expresan una cualidad del individuo, tales como el sexo, el nombre, la especie… . En R, guardaremos las listas de datos cualitativos en vectores (habitualmente, de palabras), o en factores si vamos a usarlos para clasificar individuos.
Datos ordinales. Son datos similares a los cualitativos, con la única diferencia de que se pueden ordenar de manera natural. Por ejemplo, los niveles de calidad ambiental de un ecosistema (malo, regular, normal, bueno, muy bueno) o las calificaciones en un examen (suspenso, aprobado, notable, sobresaliente) son datos ordinales. En cambio, no se pueden ordenar de manera significativa los sexos o las especies de los individuos. En R, guardaremos las listas de datos ordinales en factores ordenados.
Datos cuantitativos. Son datos que se refieren a medidas, tales como edades, longitudes, pesos, tiempos, números de individuos, etc. En R, guardaremos las listas de datos cuantitativos en vectores de números.
El análisis, tanto descriptivo como inferencial, de un conjunto de datos es diferente según su tipo. Así, para datos cualitativos sólo tiene interés estudiar y representar las frecuencias con que aparecen sus diferentes valores, mientras que el análisis de datos cuantitativos suele involucrar el cálculo de medidas estadísticas que evalúen numéricamente sus propiedades.
6.1 Estructura de un data frame
La instalación básica de R lleva predefinidos algunos objetos de datos.
Podemos echarles un vistazo entrando la instrucción data(), que mostrará en la ventana de ficheros la lista de los objetos de datos a los que tenemos acceso en la sesión actual de R (los que lleva la instalación básica de R y los que aporten los paquetes que tengamos cargados). Estos objetos no aparecen cuando hacemos ls() ni se borran con rm(list=ls()), y podemos pedir información sobre cada uno de ellos en la ventana de Ayuda. Al final de la lista que obtenemos con data() se nos indica que si entramos
data(package=.packages(all.available=TRUE))obtendremos la lista de todos los objetos de datos a los que tenemos acceso, incluyendo los de los paquetes instalados pero no cargados en la sesión actual.
Una de las tablas de datos más populares que lleva R es el llamado iris data set, que contiene la longitud y la anchura de los pétalos y sépalos y la especie de 150 flores iris. El famoso estadístico Sir R. A. Fisher usó este conjunto de datos en su artículo “The Use of Multiple Measurements in Taxonomic Problems” (publicado en Annals of Eugenics 7 (1936), pp. 179-188, y que podéis descargar de http://digital.library.adelaide.edu.au/coll/special//fisher/138.pdf). En R, este conjunto de datos de flores iris está recogido en el data frame iris. Para más información sobre estos datos, podéis leer su entrada en la Wikipedia y para más información específica sobre la tabla iris de R, podéis consultar la Ayuda de iris.
En esta sección vamos a trabajar con esta tabla de datos, así que lo primero que haremos será copiarla en un nuevo data frame que llamaremos d.f; de este modo trabajaremos sobre la copia d.f y tendremos acceso al data frame iris original si necesitamos volver a él. De todas formas, si trabajamos directamente sobre iris y lo echamos a perder, lo podemos recuperar ejecutando la instrucción data(iris), que devolverá a este objeto su contenido original.
d.f=irisSi pidiéramos ahora a R que nos mostrase el objeto d.f mediante
d.fobtendríamos el contenido del data frame en la consola: una larga lista de datos formada por las 150 filas de la tabla; naturalmente, no vamos a copiar aquí esta salida. Para echarle un vistazo al data frame, y así poder entender su estructura y conocer los nombres de sus variables, os recomendamos usar la instrucción
View(d.f)que lo mostrará en la ventana superior izquierda de RStudio: véase la Figura 6.1.
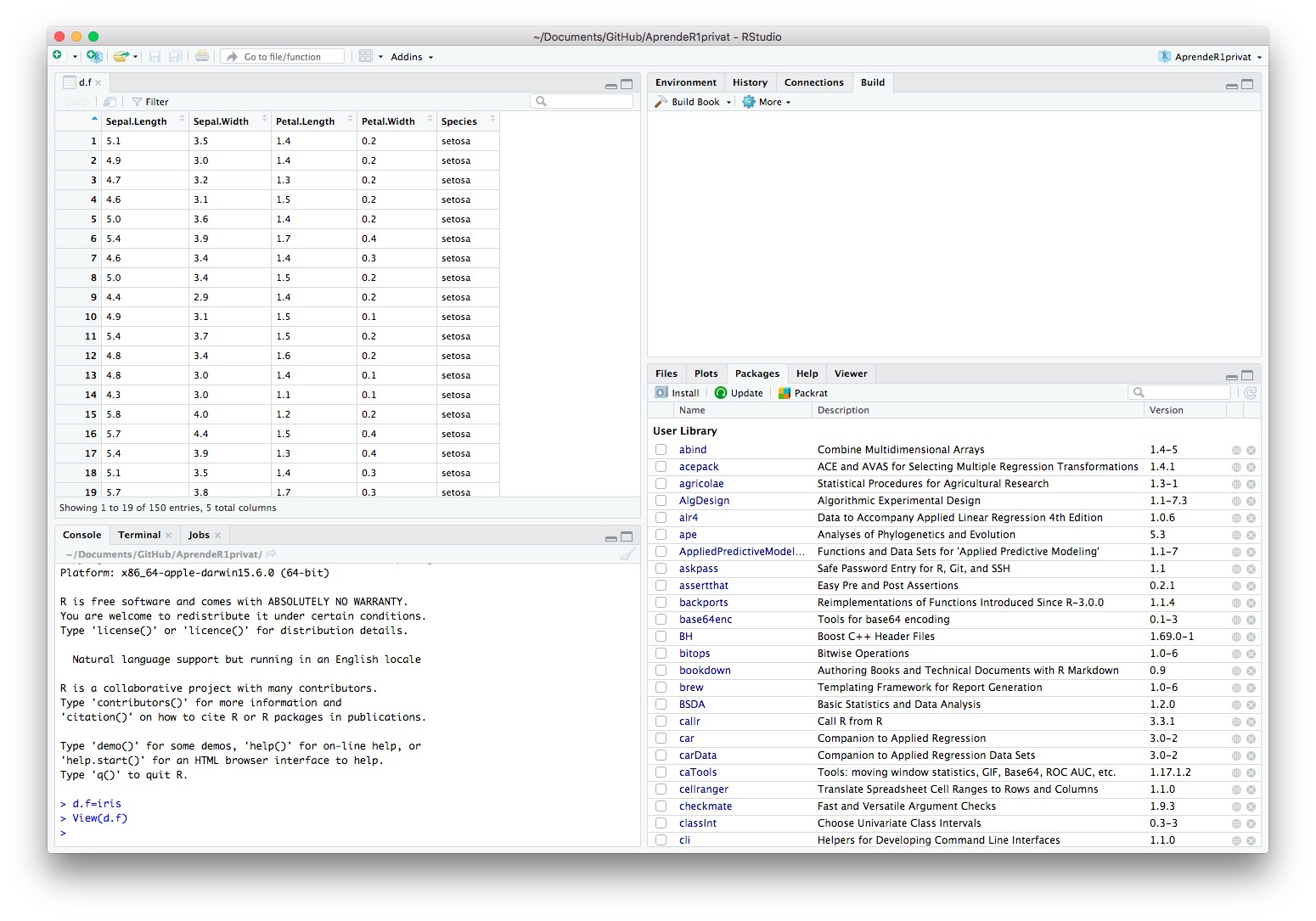
Figura 6.1: Vista de un data frame con View.
También podemos consultar en la consola las primeras filas del data frame, aplicando la función head al data frame y al número de filas que queremos que muestre; su valor por defecto es 6.
head(d.f, 5) #Las primeras 5 filas
#> Sepal.Length Sepal.Width Petal.Length Petal.Width Species
#> 1 5.1 3.5 1.4 0.2 setosa
#> 2 4.9 3.0 1.4 0.2 setosa
#> 3 4.7 3.2 1.3 0.2 setosa
#> 4 4.6 3.1 1.5 0.2 setosa
#> 5 5.0 3.6 1.4 0.2 setosaLa función tail, con una estructura similar a head, nos muestra la “cola” de la tabla.
tail(d.f, 5) #Las últimas 5 filas
#> Sepal.Length Sepal.Width Petal.Length Petal.Width Species
#> 146 6.7 3.0 5.2 2.3 virginica
#> 147 6.3 2.5 5.0 1.9 virginica
#> 148 6.5 3.0 5.2 2.0 virginica
#> 149 6.2 3.4 5.4 2.3 virginica
#> 150 5.9 3.0 5.1 1.8 virginicaObservad que las columnas del data frame tienen nombres (R los llama names) y las filas, que corresponden a individuos, tienen como identificador (R llama a estos identificadores rownames) un número natural correlativo que va del 1 al 150. Podemos comprobar también que, en cada fila, la columna Species contiene una palabra que describe la especie de la flor, mientras que las otras cuatro columnas contienen números que corresponden a medidas.
Como veremos, la columna Species no es un vector propiamente dicho, sino un factor (con niveles las tres especies de iris: setosa, versicolor y virginica), lo que nos facilita el uso de esta variable para clasificar las flores.
Si queremos conocer la estructura global de un data frame, le podemos aplicar la función str.
str(d.f)
#> 'data.frame': 150 obs. of 5 variables:
#> $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
#> $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
#> $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
#> $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
#> $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...El resultado de esta instrucción nos muestra la estructura del objeto d.f. Nos dice que es un data frame formado por 150 observaciones (filas) de 5 variables (columnas), y de cada variable nos da su nombre (precedido de un signo $) y su tipo de datos: las cuatro primeras son variables cuantitativas, formadas por vectores numéricos (num), y la quinta es una variable cualitativa, un factor con (w/, de with) 3 niveles que corresponden a las especies. Además, nos muestra los primeros valores de cada variable: en el caso del factor, los unos significan setosa, su primer nivel por orden alfabético.
Las funciones siguientes nos permiten obtener los nombres de las variables, los identificadores de las filas y las dimensiones de un data frame:
names: Produce un vector con los nombres de las columnas.rownames: Produce un vector con los identificadores de las filas.dimnames: Produce unalistformada por dos vectores: el de los identificadores de las filas y el de los nombres de las columnas.dim: Produce un vector con el número de filas y el número de columnas.
Veamos sus valores sobre nuestro data frame:
names(d.f)
#> [1] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width" "Species"
rownames(d.f)
#> [1] "1" "2" "3" "4" "5" "6" "7" "8" "9" "10" "11" "12"
#> [13] "13" "14" "15" "16" "17" "18" "19" "20" "21" "22" "23" "24"
#> [25] "25" "26" "27" "28" "29" "30" "31" "32" "33" "34" "35" "36"
#> [37] "37" "38" "39" "40" "41" "42" "43" "44" "45" "46" "47" "48"
#> [49] "49" "50" "51" "52" "53" "54" "55" "56" "57" "58" "59" "60"
#> [61] "61" "62" "63" "64" "65" "66" "67" "68" "69" "70" "71" "72"
#> [73] "73" "74" "75" "76" "77" "78" "79" "80" "81" "82" "83" "84"
#> [85] "85" "86" "87" "88" "89" "90" "91" "92" "93" "94" "95" "96"
#> [97] "97" "98" "99" "100" "101" "102" "103" "104" "105" "106" "107" "108"
#> [109] "109" "110" "111" "112" "113" "114" "115" "116" "117" "118" "119" "120"
#> [121] "121" "122" "123" "124" "125" "126" "127" "128" "129" "130" "131" "132"
#> [133] "133" "134" "135" "136" "137" "138" "139" "140" "141" "142" "143" "144"
#> [145] "145" "146" "147" "148" "149" "150"
dimnames(d.f)
#> [[1]]
#> [1] "1" "2" "3" "4" "5" "6" "7" "8" "9" "10" "11" "12"
#> [13] "13" "14" "15" "16" "17" "18" "19" "20" "21" "22" "23" "24"
#> [25] "25" "26" "27" "28" "29" "30" "31" "32" "33" "34" "35" "36"
#> [37] "37" "38" "39" "40" "41" "42" "43" "44" "45" "46" "47" "48"
#> [49] "49" "50" "51" "52" "53" "54" "55" "56" "57" "58" "59" "60"
#> [61] "61" "62" "63" "64" "65" "66" "67" "68" "69" "70" "71" "72"
#> [73] "73" "74" "75" "76" "77" "78" "79" "80" "81" "82" "83" "84"
#> [85] "85" "86" "87" "88" "89" "90" "91" "92" "93" "94" "95" "96"
#> [97] "97" "98" "99" "100" "101" "102" "103" "104" "105" "106" "107" "108"
#> [109] "109" "110" "111" "112" "113" "114" "115" "116" "117" "118" "119" "120"
#> [121] "121" "122" "123" "124" "125" "126" "127" "128" "129" "130" "131" "132"
#> [133] "133" "134" "135" "136" "137" "138" "139" "140" "141" "142" "143" "144"
#> [145] "145" "146" "147" "148" "149" "150"
#>
#> [[2]]
#> [1] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width" "Species"
dim(d.f)
#> [1] 150 5Fijaos en que el resultado de rownames son los identificadores de las filas entre comillas, es decir, considerados como palabras; R entiende siempre que estos identificadores son palabras, aunque, como en este caso, sean números.
Recordaréis que se puede obtener el valor de una componente de una list añadiendo al nombre de esta última el sufijo formado por el signo $ seguido del nombre de la componente. De manera similar, para obtener una columna concreta de un data frame basta añadir al nombre de este último el sufijo formado por el signo$seguido del nombre de la variable; el resultado será un vector o un factor, según cómo esté definida la columna dentro del data frame.
d.f$Sepal.Length[1:30]
#> [1] 5.1 4.9 4.7 4.6 5.0 5.4 4.6 5.0 4.4 4.9 5.4 4.8 4.8 4.3 5.8 5.7 5.4 5.1 5.7
#> [20] 5.1 5.4 5.1 4.6 5.1 4.8 5.0 5.0 5.2 5.2 4.7
d.f$Species[1:30]
#> [1] setosa setosa setosa setosa setosa setosa setosa setosa setosa setosa
#> [11] setosa setosa setosa setosa setosa setosa setosa setosa setosa setosa
#> [21] setosa setosa setosa setosa setosa setosa setosa setosa setosa setosa
#> Levels: setosa versicolor virginicaComo también pasaba con las componentes de las list, las variables de un data frame son internas, no están definidas en el entorno global de trabajo de R.
Sepal.Length
#> Error in eval(expr, envir, enclos): objeto 'Sepal.Length' no encontradoEn la Sección 6.8 explicaremos cómo podemos declarar las variables internas de un data frame como variables globales, y así poder usarlas directamente por su nombre, sin tener que añadirles delante el nombre del data frame y el $.
Los data frames comparten con las matrices el uso de los corchetes para extraer trozos por filas y columnas. Los resultados que se obtienen son de nuevo data frames, y tanto los nombres de las columnas como los identificadores de las filas se heredan del data frame original. Podemos comprobarlo en los siguientes ejemplos.
- La subtabla formada por las 5 primeras filas de
d.f:
d.f[1:5, ]
#> Sepal.Length Sepal.Width Petal.Length Petal.Width Species
#> 1 5.1 3.5 1.4 0.2 setosa
#> 2 4.9 3.0 1.4 0.2 setosa
#> 3 4.7 3.2 1.3 0.2 setosa
#> 4 4.6 3.1 1.5 0.2 setosa
#> 5 5.0 3.6 1.4 0.2 setosa- La subtabla formada por las 3 primeras columnas de las 5 primeras filas de
d.f:
d.f[1:5, 1:3]
#> Sepal.Length Sepal.Width Petal.Length
#> 1 5.1 3.5 1.4
#> 2 4.9 3.0 1.4
#> 3 4.7 3.2 1.3
#> 4 4.6 3.1 1.5
#> 5 5.0 3.6 1.4- La subtabla formada por las filas correspondientes a las flores de especie virginica cuya longitud del sépalo es mayor que 7:
d.f[d.f$Species=="virginica" & d.f$Sepal.Length>7, ]
#> Sepal.Length Sepal.Width Petal.Length Petal.Width Species
#> 103 7.1 3.0 5.9 2.1 virginica
#> 106 7.6 3.0 6.6 2.1 virginica
#> 108 7.3 2.9 6.3 1.8 virginica
#> 110 7.2 3.6 6.1 2.5 virginica
#> 118 7.7 3.8 6.7 2.2 virginica
#> 119 7.7 2.6 6.9 2.3 virginica
#> 123 7.7 2.8 6.7 2.0 virginica
#> 126 7.2 3.2 6.0 1.8 virginica
#> 130 7.2 3.0 5.8 1.6 virginica
#> 131 7.4 2.8 6.1 1.9 virginica
#> 132 7.9 3.8 6.4 2.0 virginica
#> 136 7.7 3.0 6.1 2.3 virginica- La subtabla formada por las 4 primeras filas de la subtabla anterior:
d.f[d.f$Species=="virginica" & d.f$Sepal.Length>7, ][1:4, ]
#> Sepal.Length Sepal.Width Petal.Length Petal.Width Species
#> 103 7.1 3.0 5.9 2.1 virginica
#> 106 7.6 3.0 6.6 2.1 virginica
#> 108 7.3 2.9 6.3 1.8 virginica
#> 110 7.2 3.6 6.1 2.5 virginicaEn todos los casos, obtenemos subtablas del data frame d.f con sus filas y columnas determinadas por las expresiones entre corchetes.
6.2 Cómo importar y exportar data frames
R dispone de varias instrucciones que permiten importar ficheros externos como objetos de datos; por ejemplo, ya vimos en la Sección 4.1 la función scan, que permitía definir un vector con el contenido de un fichero. En esta sección explicaremos cómo podemos definir un data frame a partir de una tabla de datos contenida en un fichero externo. Para ello, usaremos la función read.table o el menú Import Dataset de la pestaña Environment de la ventana superior derecha de RStudio.
Empecemos con la función read.table. Para crear un data frame a partir de un fichero de texto simple que contenga una tabla de datos, podemos aplicar esta función al nombre del fichero, si está en el directorio de trabajo de R, o a su url, si está en Internet; en ambos casos, entre comillas. Esta función read.table tiene algunos parámetros que evitan errores en la creación del data frame:
sep. Como ya ocurría con la funciónscan, la funciónread.tableentiende por defecto que las separaciones entre columnas en el fichero están marcadas por espacios en blanco. Si esta separación está marcada por comas, signos de punto y coma, tabuladores o de cualquier otra manera, hay que especificarlo con este parámetro. Por ejemplo, tenemos que especificarsep=","para indicar que las separaciones entre columnas son comas,sep=";"para indicar que las separaciones entre columnas son signos de punto y coma, ysep="\t"para indicar que las separaciones entre columnas son tabuladores.header. Si la tabla que importamos tiene una primera fila con los nombres de las columnas, es conveniente especificarlo con el parámetroheader=TRUE; en caso contrario, y según cómo esté formateado el fichero, puede que R entienda la fila de los nombres de las variables como la primera fila de observaciones. Si, en cambio, las columnas de la tabla que importamos no tienen nombre, no hace falta especificar el parámetroheader(o usarheader=FALSE, que es su valor por defecto), y después ya pondremos nombres a las variables con la funciónnames(véase la Sección 6.4).dec. Como ya explicamos en la funciónscan, podemos usar el parámetrodecpara especificar el separador decimal, si no es un punto.stringsAsFactors. Por defecto,read.tabletransforma en factores las columnas de palabras de la tabla que importa. Para impedir esta transformación, de manera que los vectores de palabras se importen como tales, podemos usarstringsAsFactors=FALSE.encoding. Como también ya explicamos en el contexto de la funciónscan, este parámetro sirve para indicar la codificación de alfabeto del fichero que se va a importar, y se ha de usar si dicho fichero contiene palabras con letras acentuadas o caracteres especiales y su codificación no coincide con la que espera nuestro ordenador: lo notaremos porque si importamos el fichero sin especificar este parámetro, los acentos se importan mal. Como los ficheros externos que usamos en este curso están codificados en utf8, los usuarios de Windows de vez en cuando tendrán que usarencoding="latin1". Avisaremos cada vez que sea necesario.
Para otros parámetros, podéis consultar la Ayuda de read.table.
Vamos a ilustrar el uso de esta función con los ficheros los dos urls siguientes:
Ambos ficheros son básicamente la misma tabla de datos, que recoge, para algunos estudiantes de primer curso de la UIB de hace unos años, el grado en el que estaban matriculados (Biología, BL, o Bioquímica, BQ), su número de hermanos y la nota que obtuvieron en un determinado examen; la diferencia es que, en el primero, “NotaHermanos.txt”, las columnas están separadas por espacios en blanco, y en el segundo, “NotaHermanosc.txt”, por comas. Ambos ficheros contienen una primera fila con los nombres de las variables. Están codificados en utf8, pero como no contienen letras acentuadas ni caracteres especiales, no tenéis que preocuparos por su codificación.
Para crear un data frame llamado NH1 a partir del fichero NotaHermanos.txt, basta aplicar la función read.table a su url entre comillas y especificar header=TRUE. Naturalmente, para poder importar un fichero de Internet, hay que estar conectados a Internet, el servidor que aloja el fichero tiene que funcionar, y además no tiene que requerir una palabra clave para acceder.
NH1=read.table("http://aprender.uib.es/Rdir/NotaHermanos.txt", header=TRUE)Es una buena costumbre, una vez definido un data frame a partir de un fichero externo, comprobar que se ha importado bien. Como una tabla de datos puede tener muchas filas, verla entera en la consola puede ser poco práctico; lo mejor es usar las funciones str y View o head. Os aconsejamos que, siempre que vayáis a trabajar con un data frame, le echéis antes un vistazo con estas funciones para comprobar su estructura, los nombres de sus variables, los tipos de sus datos, etc. Esto es especialmente importante cuando se trata de data frames importados, porque si no hemos especificado los parámetros adecuados, puede que en el proceso de lectura y definición del data frame se hayan perdido los nombres de las variables o la estructura de columnas.
head(NH1)
#> Grado Hermanos Nota
#> 1 BQ 1 93
#> 2 BL 1 35
#> 3 BL 1 55
#> 4 BL 1 77
#> 5 BL 2 81
#> 6 BQ 0 86
str(NH1)
#> 'data.frame': 87 obs. of 3 variables:
#> $ Grado : chr "BQ" "BL" "BL" "BL" ...
#> $ Hermanos: int 1 1 1 1 2 0 1 1 1 1 ...
#> $ Nota : int 93 35 55 77 81 86 97 40 16 31 ...Si no hubiéramos especificado header=TRUE, el formato del data frame resultante no habría sido el adecuado.
NH1.mal=read.table("http://aprender.uib.es/Rdir/NotaHermanos.txt")
str(NH1.mal)
#> 'data.frame': 88 obs. of 3 variables:
#> $ V1: chr "Grado" "BQ" "BL" "BL" ...
#> $ V2: chr "Hermanos" "1" "1" "1" ...
#> $ V3: chr "Nota" "93" "35" "55" ...Sin el parámetro header=TRUE, R ha entendido que Grado, Hermanos y Nota son elementos de sus columnas respectivas, y no sus nombres; en consecuencia, por un lado, ha llamado por defecto V1, V2 y V3 a las columnas del data frame, y por otro, como Grado, Hermanos y Nota son palabras y los datos de cada variable de un data frame han de ser del mismo tipo, ha considerado que todas las entradas de cada columna eran palabras. Finalmente, como, al crear un data frame, R transforma los vectores de palabras en factores si no especificamos lo contrario, hemos obtenido un data frame formado por tres factores, y hemos perdido la información numérica que contenían las columnas con el número de hermanos y la nota.
Para importar el fichero NotaHermanosc.txt, donde las columnas están separadas por comas, hay que usar el parámetro sep=",".
NH2=read.table("http://aprender.uib.es/Rdir/NotaHermanosc.txt", header=TRUE, sep=",")
str(NH2)
#> 'data.frame': 87 obs. of 3 variables:
#> $ Grado : chr "BQ" "BL" "BL" "BL" ...
#> $ Hermanos: int 1 1 1 1 2 0 1 1 1 1 ...
#> $ Nota : int 93 35 55 77 81 86 97 40 16 31 ...Sin este parámetro, R hubiera entendido cada fila como una sola palabra.
NH2.mal=read.table("http://aprender.uib.es/Rdir/NotaHermanosc.txt", header=TRUE)
str(NH2.mal)
#> 'data.frame': 87 obs. of 1 variable:
#> $ Grado.Hermanos.Nota: chr "BQ,1,93" "BL,1,35" "BL,1,55" "BL,1,77" ...Veamos el efecto de stringsAsFactors=FALSE:
NH3=read.table("http://aprender.uib.es/Rdir/NotaHermanosc.txt", header=TRUE,
sep=",", stringsAsFactors=FALSE)
str(NH3)
#> 'data.frame': 87 obs. of 3 variables:
#> $ Grado : chr "BQ" "BL" "BL" "BL" ...
#> $ Hermanos: int 1 1 1 1 2 0 1 1 1 1 ...
#> $ Nota : int 93 35 55 77 81 86 97 40 16 31 ...La variable Grado se ha importado como un vector de palabras: lo indica el chr, de character, en la fila correspondiente del resultado de str.
Para importar versiones locales de estos ficheros, tendríamos que usar las mismas instrucciones, cambiando el url por el nombre del fichero, siempre entre comillas. Por ejemplo, si disponemos de una copia del fichero NotaHermanos.txt en nuestro escritorio de trabajo, lo podemos cargar con la instrucción:
NH4=read.table("NotaHermanos.txt", header=TRUE)
str(NH4)
#> 'data.frame': 87 obs. of 3 variables:
#> $ Grado : chr "BQ" "BL" "BL" "BL" ...
#> $ Hermanos: int 1 1 1 1 2 0 1 1 1 1 ...
#> $ Nota : int 93 35 55 77 81 86 97 40 16 31 ...Para importar tablas de datos también se puede usar el menú Import Dataset de la pestaña Environment, y de hecho esta es la opción más aconsejable cuando usamos R de manera interactiva. Este menú dispone de varias opciones, según el tipo de fichero (texto simple, Excel, SPSS …) y según que esté en el ordenador o en un url (si entráis un url pulsad a continuación el botón Update para que se conecte). Según la opción que escojáis, puede ser que tengáis que instalar algún paquete, como readr o readxl, ya os avisará RStudio con un mensaje de error. En cada caso, se os presentarán diversas opciones que os permitirán, por ejemplo, poner nombre al data frame (en el campo Name), indicar si tiene una primera fila con los nombres de las variables (seleccionando Yes en el campo Heading o marcando el botón First Row as Names, según la opción del menú que hayáis elegido), especificar el signo que separa las columnas (Separator o Delimiter, de nuevo según la opción del menú que hayáis elegido) y cómo aparecen identificados los valores NA en el fichero, etc. La ventaja de estos menús es que podéis ver el aspecto del data frame que crearéis con nuestras elecciones. Su inconveniente es que, si queréis que vuestro análisis de datos sea reproducible por otras personas, es necesario incluir en el documento que publiquéis la instrucción usada para cargar los datos. Por suerte, en la mayoría de opciones de este menú podéis ver las instrucciones que se usan y podéis copiarlas. En las Figuras 6.2 y 6.3 mostramos las ventanas que aparecen usando en el menú Import Dataset las opciones From Text (base)… (para importar con read.table un fichero local) y From Text (readr)… (para importar con funciones del paquete readr ficheros locales o en línea) para importar el fichero “NotaHemanosc.txt”. En esta última, observad que en el campo inferior derecho (Code Preview) aparecen las instrucciones que usará R y que podéis copiar (no copiéis el View, naturalmente).
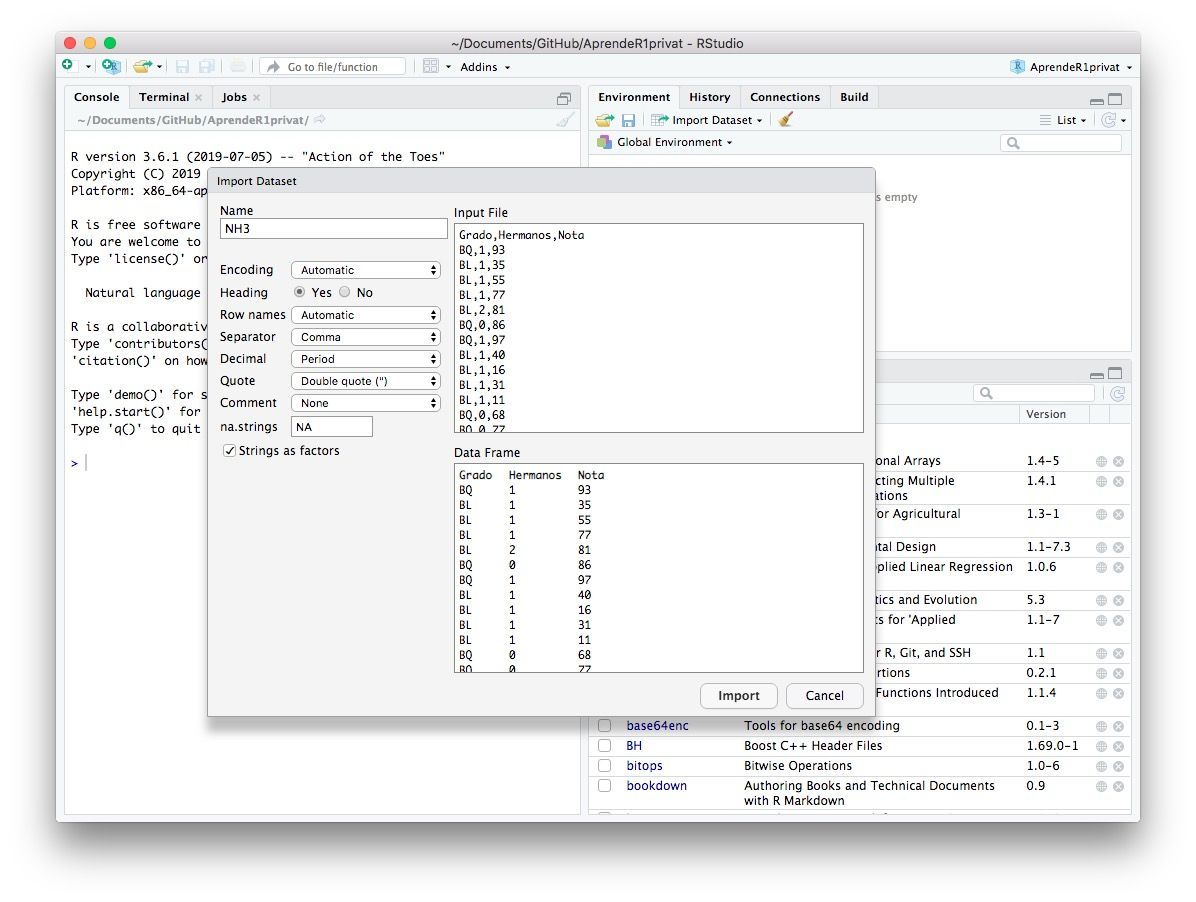
Figura 6.2: Importador de tablas de datos del Rstudio usando From Text (base)….
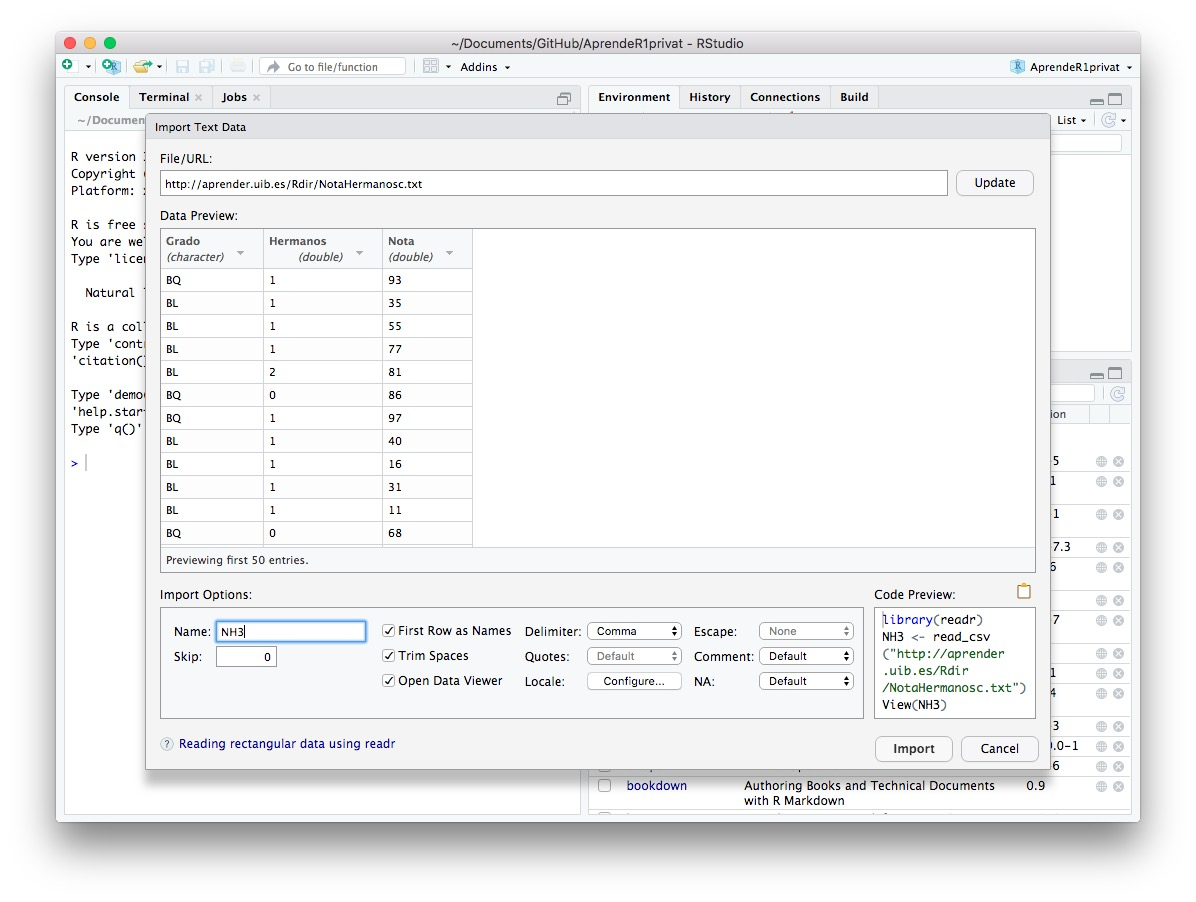
Figura 6.3: Importador de tablas de datos del Rstudio usando From Text (readr)….
Podemos exportar un data frame a un fichero usando la función write.table. Su sintaxis básica es
write.table(data frame, file="nombre del fichero")Esta función crea un fichero, en el directorio de trabajo de R, que contiene el data frame que hemos especificado en el argumento, y lo llama el nombre que hemos especificado entrecomillado en file. Además, por ejemplo, podemos usar el parámetro sep para indicar el signo de separación de columnas en el fichero que creemos.
Así, por ejemplo, vamos a crear un data frame llamado A con un trozo de iris.
A=iris[1:5, 1:4]
A
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 1 5.1 3.5 1.4 0.2
#> 2 4.9 3.0 1.4 0.2
#> 3 4.7 3.2 1.3 0.2
#> 4 4.6 3.1 1.5 0.2
#> 5 5.0 3.6 1.4 0.2Para guardar este data frame A en un fichero llamado trozoiris.txt donde las columnas aparezcan separadas con signos de punto y coma, entramos:
write.table(A, file="trozoiris.txt", sep=";")Y ahora podemos volver a importar este fichero con un read.table adecuado:
B=read.table("trozoiris.txt", header=TRUE, sep=";")
B
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 1 5.1 3.5 1.4 0.2
#> 2 4.9 3.0 1.4 0.2
#> 3 4.7 3.2 1.3 0.2
#> 4 4.6 3.1 1.5 0.2
#> 5 5.0 3.6 1.4 0.2Para terminar, una última instrucción para importar tablas, basada en funciones del paquete readr. Podéis seleccionar y copiar toda el contenido de una tabla de datos en el portapapeles con la combinación de teclas que corresponda en vuestro sistema operativo y “pegarlo” en un data frame con la siguiente instrucción (tras cargar primero el paquete readr si es necesario):
Nombre=read_delim(clipboard(),delim=...)donde Nombre refiere al nombre que queréis dar al data frame y en el parámetro delim tenéis que especificar el separador de columnas de manera similar a cómo usamos sep en read.table; la función clipboard() de readr devuelve el contenido del portapapeles. Leed la Ayuda de la función read_delim para conocer otros parámetros que podéis especificar.
6.3 Cómo crear data frames
Además de trabajar con data frames importados o predefinidos, también podemos crearlos; para ello, organizaremos en forma de tabla algunos vectores, cada uno de los cuales contendrá las observaciones de una variable para un conjunto de individuos o casos, y de manera que los datos en todos estos vectores estén en el mismo orden de los individuos: es decir, que la primera entrada de cada vector corresponda a un mismo individuo, la segunda entrada de cada vector corresponda a otro mismo individuo, y así sucesivamente.
Para construir un data frame a partir de unos vectores, se usa la función data.frame aplicada a los vectores en el orden en el que queramos disponer las columnas de la tabla;
de esta manera, las variables tomarán los nombres de los vectores. Estos nombres también se pueden especificar en el argumento de la función data.frame, entrando cada columna con una construcción de la forma
Nombre de la variable=Vector con el contenido de la variableVamos a ilustrar esta función con un ejemplo sencillo, que arrastraremos durante buena parte de lo que queda de lección. Vamos a construir un data frame que contenga algunos datos sobre estudiantes; concretamente, este data frame tendrá tres variables: una primera columna con el sexo del estudiante, la segunda columna con su edad en años y la tercera con su número de hermanos. Como ejemplo de la construcción anterior, a los vectores conteniendo las dos primeras variables les pondremos de entrada nombres razonables, que mantendremos en el data frame, y al vector con los números de hermanos lo llamaremos X y le asignaremos el nombre Hermanos al crear el data frame.
Sexo=c("Hombre","Hombre","Mujer","Hombre","Hombre","Hombre","Mujer")
Edad=c(17,18,20,18,18,18,19)
X=c(2,0,0,1,1,1,0)
d.f1=data.frame(Sexo, Edad, Hermanos=X)
d.f1
#> Sexo Edad Hermanos
#> 1 Hombre 17 2
#> 2 Hombre 18 0
#> 3 Mujer 20 0
#> 4 Hombre 18 1
#> 5 Hombre 18 1
#> 6 Hombre 18 1
#> 7 Mujer 19 0Puesto que iremos modificando este data frame, vamos a guardar una copia de seguridad (un backup) en d.f1bk para poder recuperar su estado original si lo necesitamos.
d.f1bk=d.f1Es muy prudente y recomendable que guardéis siempre copias de seguridad de los data frames que generéis si vais a manipularlos, porque hay cambios en un data frame que son irreversibles y, por lo tanto, si os arrepentís de haber hecho algún cambio, bien podría ser que no lo pudierais deshacer.
Vamos a consultar algunas características del data frame d.f1.
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : num 17 18 20 18 18 18 19
#> $ Hermanos: num 2 0 0 1 1 1 0
rownames(d.f1)
#> [1] "1" "2" "3" "4" "5" "6" "7"Vemos que la primera variable es un factor con dos niveles, y las otras dos son vectores numéricos (num). Vemos también que R ha asignado como identificadores de las filas los números del 1 al 7, pero los considera palabras.
La función data.frame dispone de algunos parámetros que permiten ajustar a nuestras necesidades el data frame que creamos. Los más útiles son los siguientes:
row.names. Sirve para especificar los identificadores de las filas.stringsAsFactors. Por defecto,data.frameconvierte los vectores de palabras en factores. ConstringsAsFactors=FALSE, imponemos que los vectores de palabras se mantengan como tales en el data frame
Veamos un ejemplo de aplicación de estos dos parámetros.
d.f2=data.frame(Sexo, Edad, Hermanos=X, stringsAsFactors=FALSE,
row.names=c("E1","E2","E3","E4","E5","E6","E7"))
d.f2
#> Sexo Edad Hermanos
#> E1 Hombre 17 2
#> E2 Hombre 18 0
#> E3 Mujer 20 0
#> E4 Hombre 18 1
#> E5 Hombre 18 1
#> E6 Hombre 18 1
#> E7 Mujer 19 0
str(d.f2)
#> 'data.frame': 7 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : num 17 18 20 18 18 18 19
#> $ Hermanos: num 2 0 0 1 1 1 0
d.f2$Sexo
#> [1] "Hombre" "Hombre" "Mujer" "Hombre" "Hombre" "Hombre" "Mujer"Como vemos, el efecto de stringsAsFactors=FALSE ha sido que la variable Sexo es ahora un vector de palabras, y el del parámetro row.names ha sido llamar E1,E2,...,E7 a las filas.
Si hemos asignado identificadores a las filas, podemos usarlos para extraer subtablas del data frame (aunque también podemos seguir usando los números de las filas). Recordad que, si usáis los identificadores, como son palabras, hay que escribirlos entre comillas.
d.f2[c("E1","E2"), ] #Subtabla con las filas E1 y E2
#> Sexo Edad Hermanos
#> E1 Hombre 17 2
#> E2 Hombre 18 0Para crear un data frame a partir de cero, entrando los datos en una tabla, lo más práctico es usar un programa externo, por ejemplo Open Office, para crear la tabla de datos, y luego importar la tabla con el menú Import Dataset tal y como explicábamos en la sección anterior.
6.4 Cómo modificar un data frame
En esta sección veremos la manera de modificar un data frame una vez creado o importado.
Para cambiar los nombres de las variables, podemos usar la instrucción
names(data frame)=vector con los nombres de las variablesVolvamos al d.f1. Tras recordar su estructura, cambiaremos los nombres de sus variables, sustituyéndolos por sus iniciales, y volveremos a consultar su estructura para ver cómo han cambiado estos nombres.
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : num 17 18 20 18 18 18 19
#> $ Hermanos: num 2 0 0 1 1 1 0
names(d.f1)=c("S","E","H")
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ S: chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ E: num 17 18 20 18 18 18 19
#> $ H: num 2 0 0 1 1 1 0Si sólo queremos cambiar el nombre de algunas variables, basta redefinir el trozo correspondiente del vector names. Así, si en la nueva copia de d.f1 quisiéramos volver a cambiar el nombre de la variable E por Edad, podríamos usar una de las dos instrucciones siguientes:
names(d.f1)[2]="Edad"o
names(d.f1)[names(d.f1)=="E"]="Edad"Con la primera, cambiaríamos el nombre de la segunda variable por Edad; con la segunda, cambiaríamos el nombre de la variable llamada E por Edad. En los dos casos, la estructura del data frame resultante es la siguiente:
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ S : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad: num 17 18 20 18 18 18 19
#> $ H : num 2 0 0 1 1 1 0Para modificar los identificadores de las filas, podemos usar la instrucción
rownames(data frame)=vector con los nombres de las filasComo cada identificador ha de determinar el individuo al que corresponde la fila, conviene que estos identificadores sean todos diferentes.
rownames(d.f1)=paste("Alumno", 1:7, sep="")
d.f1
#> S Edad H
#> Alumno1 Hombre 17 2
#> Alumno2 Hombre 18 0
#> Alumno3 Mujer 20 0
#> Alumno4 Hombre 18 1
#> Alumno5 Hombre 18 1
#> Alumno6 Hombre 18 1
#> Alumno7 Mujer 19 0Como podéis deducir de su efecto, la función paste pega vectores, entrada a entrada, usando como separador el valor del parámetro sep. El separador por defecto es un espacio en blanco, nosotros hemos optado por no usar nada para separar la palabra “Alumno” del número, y por eso hemos especificado sep="" (entre las comillas no hay nada, ni siquiera un espacio en blanco). Fiajos en que si en lugar de pegar un vector pegamos un elemento, éste se entiende como un vector constante formado por el número adecuado de copias.
Podemos modificar los nombres de las filas y de las columnas simultáneamente con la instrucción
dimnames(dataframe)=list(vector con los nombres de las filas, vector con los nombres de las columnas)Por ejemplo, vamos a cambiar de golpe en d.f1 los identificadores de las filas, para que ahora sean números romanos, y los nombres de las variables, para que pasen a ser “Var1”, “Var2” y “Var3”:
dimnames(d.f1)=list(c("I","II","III","IV","V","VI","VII"), c("Var1","Var2","Var3"))
d.f1
#> Var1 Var2 Var3
#> I Hombre 17 2
#> II Hombre 18 0
#> III Mujer 20 0
#> IV Hombre 18 1
#> V Hombre 18 1
#> VI Hombre 18 1
#> VII Mujer 19 0En lugar del vector con los números romanos, hubiéramos podido usar la función as.roman para traducir números arábigos a romanos:
as.roman(1:dim(d.f1)[1])
#> [1] I II III IV V VI VIIPara modificar entradas concretas de un data frame, podemos usar instrucciones similares a las que usábamos en los vectores y las matrices para modificar entradas. Por ejemplo, vamos a volver a la copia guardada de d.f1 y vamos a cambiar en ella la primera entrada de la tercera columna, es decir, la entrada (1,3), por un 3:
d.f1=d.f1bk
d.f1
#> Sexo Edad Hermanos
#> 1 Hombre 17 2
#> 2 Hombre 18 0
#> 3 Mujer 20 0
#> 4 Hombre 18 1
#> 5 Hombre 18 1
#> 6 Hombre 18 1
#> 7 Mujer 19 0Para referirnos a la entrada (1,3) de un data frame usamos la misma construcción que usábamos en las matrices:
d.f1[1,3]=3
d.f1
#> Sexo Edad Hermanos
#> 1 Hombre 17 3
#> 2 Hombre 18 0
#> 3 Mujer 20 0
#> 4 Hombre 18 1
#> 5 Hombre 18 1
#> 6 Hombre 18 1
#> 7 Mujer 19 0Otra posibilidad hubiera sido modificar directamente la variable con:
d.f1$Hermanos[1]=3Pero fijaos en que la modificamos invocándola con su “nombre compuesto” dataframe$variable; usar solo el nombre de la variable hubiera sido un error:
Hermanos[1]=3
#> Error in Hermanos[1] = 3: objeto 'Hermanos' no encontradoEn este caso hemos tenido suerte porque como la variable Hermanos no existe en el entorno global de R, hemos obtenido un mensaje de error. Pero por ejemplo con
Edad[1]=28no obtenemos ningún error, porque el vector Edad que hemos modificado existe fuera del data frame, pero no hemos modificado el data frame:
d.f1
#> Sexo Edad Hermanos
#> 1 Hombre 17 3
#> 2 Hombre 18 0
#> 3 Mujer 20 0
#> 4 Hombre 18 1
#> 5 Hombre 18 1
#> 6 Hombre 18 1
#> 7 Mujer 19 0
Edad
#> [1] 28 18 20 18 18 18 19Otra posibilidad es usar el editor de datos del que ya hemos hablado en la Lección 4. Recordaréis que, para abrir un objeto de datos con este editor, se le aplica la función fix. R abre entonces el objeto en una nueva ventana de edición. Los cambios que realicemos en un objeto con el editor de datos se guardarán cuando cerremos esta ventana. Es muy probable que para abrir data frames con el editor de datos de Rstudio tengáis que instalar algún programa auxiliar. Por ejemplo, en el caso del Mac OS X, se tiene que tener instalada la última versión de XQuartz, que será el programa en el que se abrirá el editor; el instalador de XQuartz se puede descargar de http://xquartz.macosforge.org.
Así, para realizar el cambio anterior, hubiéramos entrado
fix(d.f1)y se hubiera abierto una ventana como la que muestra la Figura 6.4, donde podríais efectuar los cambios que quisiérais.
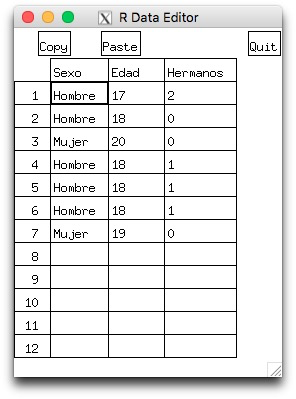
Figura 6.4: Editor de data frames de Rstudio en Mac OS X.
Veamos otro ejemplo. Volvemos de nuevo a la copia guardada de d.f1 y ahora vamos a substituir su entrada (2,2) (la segunda entrada de la segunda columna) por la palabra “Joven”:
d.f1=d.f1bk
d.f1[2,2]="Joven"
d.f1
#> Sexo Edad Hermanos
#> 1 Hombre 17 2
#> 2 Hombre Joven 0
#> 3 Mujer 20 0
#> 4 Hombre 18 1
#> 5 Hombre 18 1
#> 6 Hombre 18 1
#> 7 Mujer 19 0
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : chr "17" "Joven" "20" "18" ...
#> $ Hermanos: num 2 0 0 1 1 1 0¡Vaya! Al introducir un dato de tipo palabra en la variable Edad, toda la columna ha pasado a ser un vector de palabras: recordemos que R considera del mismo tipo de datos todas las entradas de una columna de un data frame. Y ahora ya no lo podemos arreglar volviendo a poner un número:
d.f1[2,2]=18
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : chr "17" "18" "20" "18" ...
#> $ Hermanos: num 2 0 0 1 1 1 0Parece que hemos estropeado definitivamente el data frame d.f1. Por suerte, no es así: simplemente tenemos que volver a redefinir la variable Edad como un vector numérico.
Para modificar una columna entera, hay que igualar su nombre completo (que, recordemos, se especifica añadiendo al nombre del data frame el sufijo formado por el signo $ seguido del nombre de la variable dentro del data frame) a su nuevo valor:
data frame$variable=nuevo valorEste nuevo valor puede ser el resultado de un cambio de tipo de datos aplicado a la variable. R dispone de una serie de funciones de la forma as.tipo_de_objeto, que convierten el objeto al tipo que expresa el nombre de la función. Ya hemos visto en la Sección 4.4 la función as.factor, que transforma un vector en un factor. Otras funciones de este estilo son as.character, as.integer o as.numeric, que transforman todos los datos de un objeto en palabras, números enteros o números reales, respectivamente.
Un error típico al intentar modificar la variable Edad es entrar sólo
as.numeric(d.f1$Edad)
#> [1] 17 18 20 18 18 18 19Con esta instrucción, hemos obtenido la variable Edad transformada en números, pero no hemos modificado el data frame:
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : chr "17" "18" "20" "18" ...
#> $ Hermanos: num 2 0 0 1 1 1 0Tampoco sirve entrar
Edad=as.numeric(d.f1$Edad)porque esto define un vector llamado Edad de entradas 17, 18, 20, 18, 18, 18, 19, pero sigue sin modificar la variable Edad del data frame:
Edad
#> [1] 17 18 20 18 18 18 19
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : chr "17" "18" "20" "18" ...
#> $ Hermanos: num 2 0 0 1 1 1 0La manera correcta de hacerlo es redefinir la variable del data frame con:
d.f1$Edad=as.numeric(d.f1$Edad)Comprobemos que ahora sí que ha funcionado:
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : num 17 18 20 18 18 18 19
#> $ Hermanos: num 2 0 0 1 1 1 0Veamos otros ejemplos. Vamos a convertir la variable Sexo en un vector de palabras:
d.f1$Sexo=as.character(d.f1$Sexo)
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : num 17 18 20 18 18 18 19
#> $ Hermanos: num 2 0 0 1 1 1 0Y ahora vamos a convertir la variable Hermanos en un factor ordenado:
d.f1$Hermanos=ordered(d.f1$Hermanos, levels=c(0,1,2))
str(d.f1)
#> 'data.frame': 7 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : num 17 18 20 18 18 18 19
#> $ Hermanos: Ord.factor w/ 3 levels "0"<"1"<"2": 3 1 1 2 2 2 1Naturalmente, podemos cambiar el contenido de toda una variable de otras maneras que no sea sólo modificar su tipo de datos: basta igualarla a su nuevo valor. Por ejemplo, vamos a substituir todas las entrada de la variable Edad por “Joven”:
d.f1$Edad="Joven"
d.f1
#> Sexo Edad Hermanos
#> 1 Hombre Joven 2
#> 2 Hombre Joven 0
#> 3 Mujer Joven 0
#> 4 Hombre Joven 1
#> 5 Hombre Joven 1
#> 6 Hombre Joven 1
#> 7 Mujer Joven 0Al igualar la variable Edad a la palabra “Joven”, la ha substituido por el correspondiente vector constante.
6.5 Cómo añadir filas y columnas a un data frame
La mejor manera de añadir filas a un data frame es organizándolas en un nuevo data frame con los mismos nombres de las variables, y a continuación concatenarlas al data frame usando la función rbind que ya usábamos para matrices.
d.f1=d.f1bk #Volvemos a la copia original
d.f1
#> Sexo Edad Hermanos
#> 1 Hombre 17 2
#> 2 Hombre 18 0
#> 3 Mujer 20 0
#> 4 Hombre 18 1
#> 5 Hombre 18 1
#> 6 Hombre 18 1
#> 7 Mujer 19 0
nuevas.filas=data.frame(Sexo=c("Hombre","Hombre"), Edad=c(18,18), Hermanos=c(1,2))
d.f1=rbind(d.f1, nuevas.filas)
d.f1
#> Sexo Edad Hermanos
#> 1 Hombre 17 2
#> 2 Hombre 18 0
#> 3 Mujer 20 0
#> 4 Hombre 18 1
#> 5 Hombre 18 1
#> 6 Hombre 18 1
#> 7 Mujer 19 0
#> 8 Hombre 18 1
#> 9 Hombre 18 2
str(d.f1)
#> 'data.frame': 9 obs. of 3 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : num 17 18 20 18 18 18 19 18 18
#> $ Hermanos: num 2 0 0 1 1 1 0 1 2Para añadir una variable, basta especificar el valor de la columna correspondiente; por ejemplo, vamos a añadir a d.f1 una nueva variable llamada Nueva con el producto de la edad por el número de hermanos:
d.f1$Nueva=d.f1$Edad*d.f1$Hermanos
d.f1
#> Sexo Edad Hermanos Nueva
#> 1 Hombre 17 2 34
#> 2 Hombre 18 0 0
#> 3 Mujer 20 0 0
#> 4 Hombre 18 1 18
#> 5 Hombre 18 1 18
#> 6 Hombre 18 1 18
#> 7 Mujer 19 0 0
#> 8 Hombre 18 1 18
#> 9 Hombre 18 2 36También podemos añadir columnas a la derecha de un data frame usando la función cbind; en este caso, se puede añadir directamente la columna, sin necesidad de convertirla previamente en un data frame. Por ejemplo:
d.f1=cbind(d.f1, Grado=rep("Biología", 9))
d.f1
#> Sexo Edad Hermanos Nueva Grado
#> 1 Hombre 17 2 34 Biología
#> 2 Hombre 18 0 0 Biología
#> 3 Mujer 20 0 0 Biología
#> 4 Hombre 18 1 18 Biología
#> 5 Hombre 18 1 18 Biología
#> 6 Hombre 18 1 18 Biología
#> 7 Mujer 19 0 0 Biología
#> 8 Hombre 18 1 18 Biología
#> 9 Hombre 18 2 36 Biología
str(d.f1)
#> 'data.frame': 9 obs. of 5 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Mujer" "Hombre" ...
#> $ Edad : num 17 18 20 18 18 18 19 18 18
#> $ Hermanos: num 2 0 0 1 1 1 0 1 2
#> $ Nueva : num 34 0 0 18 18 18 0 18 36
#> $ Grado : chr "Biología" "Biología" "Biología" "Biología" ...Tanto en un caso como en otro, la variable añadida ha de tener la misma longitud que las variables del data frame original: en última instancia, se trata de nuevas observaciones sobre los sujetos a los que correspondían las filas del data frame original.
Finalmente, hay que recordar que también podemos usar el editor de datos para añadir (o eliminar) filas o columnas a un data frame, abriéndolo con la función fix y manipulándolo.
6.6 Cómo seleccionar trozos de un data frame
Veamos ahora con más detalle cómo podemos extraer trozos de un data frame; el método básico es similar al que usábamos en las matrices. De hecho, para seleccionar un trozo de un data frame, podemos hacerlo exactamente como en las matrices, especificando los índices de las filas y columnas que nos interesen entre corchetes. Pero como los individuos y las variables tienen nombres, también podemos usarlos para especificar filas y columnas.
Por ejemplo, supongamos que queremos crear un nuevo data frame formado por las dos primeras filas de d.f1; podríamos hacerlo, al menos, de las tres maneras siguientes:
- Con los índices de las filas:
d.f1[1:2, ]
#> Sexo Edad Hermanos Nueva Grado
#> 1 Hombre 17 2 34 Biología
#> 2 Hombre 18 0 0 Biología- Con los identificadores de las filas:
d.f1[c("1","2"), ]
#> Sexo Edad Hermanos Nueva Grado
#> 1 Hombre 17 2 34 Biología
#> 2 Hombre 18 0 0 Biología- Con un subvector del vector de identificadores de las filas:
d.f1[rownames(d.f1)[1:2], ]
#> Sexo Edad Hermanos Nueva Grado
#> 1 Hombre 17 2 34 Biología
#> 2 Hombre 18 0 0 BiologíaPodemos usar este tipo de instrucciones para obtener las filas en un orden diferente del que presentan en el data frame original:
d.f1[c(2,3,1), ]
#> Sexo Edad Hermanos Nueva Grado
#> 2 Hombre 18 0 0 Biología
#> 3 Mujer 20 0 0 Biología
#> 1 Hombre 17 2 34 BiologíaTambién podemos seleccionar filas de un data frame mediante una condición lógica; en este caso, nos quedaremos sólo con los individuos que satisfagan esta condición. Por ejemplo, si queremos seleccionar los estudiantes de d.f1 de menos de 19 años, podemos usar la construcción siguiente:
d.f1[d.f1$Edad<19, ]
#> Sexo Edad Hermanos Nueva Grado
#> 1 Hombre 17 2 34 Biología
#> 2 Hombre 18 0 0 Biología
#> 4 Hombre 18 1 18 Biología
#> 5 Hombre 18 1 18 Biología
#> 6 Hombre 18 1 18 Biología
#> 8 Hombre 18 1 18 Biología
#> 9 Hombre 18 2 36 BiologíaEsta condición lógica puede involucrar más de una variable. Por ejemplo, para seleccionar las filas correspondientes a los estudiantes de menos de 19 años y con un solo hermano, podríamos entrar:
d.f1[d.f1$Edad<19 & d.f1$Hermanos==1, ]
#> Sexo Edad Hermanos Nueva Grado
#> 4 Hombre 18 1 18 Biología
#> 5 Hombre 18 1 18 Biología
#> 6 Hombre 18 1 18 Biología
#> 8 Hombre 18 1 18 BiologíaEn cada caso, hemos obtenido un data frame. Vamos a llamar d.f.18 al primero.
d.f.18=d.f1[d.f1$Edad<19, ]
d.f.18
#> Sexo Edad Hermanos Nueva Grado
#> 1 Hombre 17 2 34 Biología
#> 2 Hombre 18 0 0 Biología
#> 4 Hombre 18 1 18 Biología
#> 5 Hombre 18 1 18 Biología
#> 6 Hombre 18 1 18 Biología
#> 8 Hombre 18 1 18 Biología
#> 9 Hombre 18 2 36 Biología
str(d.f.18)
#> 'data.frame': 7 obs. of 5 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Hombre" "Hombre" ...
#> $ Edad : num 17 18 18 18 18 18 18
#> $ Hermanos: num 2 0 1 1 1 1 2
#> $ Nueva : num 34 0 18 18 18 18 36
#> $ Grado : chr "Biología" "Biología" "Biología" "Biología" ...Como vemos, las columnas que son factores heredan en estos sub data frames todos los niveles del factor original, aunque no aparezcan en el trozo que hemos extraído. Podemos borrar los niveles sobrantes de todos los factores redefiniendo el data frame como el resultado de aplicarle la función
droplevels.
d.f.18=droplevels(d.f.18)
str(d.f.18)
#> 'data.frame': 7 obs. of 5 variables:
#> $ Sexo : chr "Hombre" "Hombre" "Hombre" "Hombre" ...
#> $ Edad : num 17 18 18 18 18 18 18
#> $ Hermanos: num 2 0 1 1 1 1 2
#> $ Nueva : num 34 0 18 18 18 18 36
#> $ Grado : chr "Biología" "Biología" "Biología" "Biología" ...Naturalmente, el mismo tipo de construcción se puede utilizar para definir un data frame formado sólo por algunas variables de la tabla original, o incluso por sólo algunas filas y columnas. Si sólo queremos definir la subtabla quedándonos con algunas variables, basta aplicar el nombre del data frame al vector de variables entre corchetes. Contrariamente a lo que pasa con las matrices, en los data frames no hay necesidad de dejar el espacio en blanco seguido de una coma que serviría para indicar que nos referimos a columnas y no a filas. Por ejemplo, con las cuatro instrucciones siguientes obtenemos cada vez el mismo resultado, un data frame formado por las dos primeras variables de d.f1:
- Con la coma:
d.f1[ ,c(1,2)]
#> Sexo Edad
#> 1 Hombre 17
#> 2 Hombre 18
#> 3 Mujer 20
#> 4 Hombre 18
#> 5 Hombre 18
#> 6 Hombre 18
#> 7 Mujer 19
#> 8 Hombre 18
#> 9 Hombre 18- Sin la coma
d.f1[c(1,2)]
#> Sexo Edad
#> 1 Hombre 17
#> 2 Hombre 18
#> 3 Mujer 20
#> 4 Hombre 18
#> 5 Hombre 18
#> 6 Hombre 18
#> 7 Mujer 19
#> 8 Hombre 18
#> 9 Hombre 18- Eliminando las tres últimas columnas:
d.f1[-(3:5)]
#> Sexo Edad
#> 1 Hombre 17
#> 2 Hombre 18
#> 3 Mujer 20
#> 4 Hombre 18
#> 5 Hombre 18
#> 6 Hombre 18
#> 7 Mujer 19
#> 8 Hombre 18
#> 9 Hombre 18- Usando los nombres de las variables:
d.f1[c("Sexo","Edad")]
#> Sexo Edad
#> 1 Hombre 17
#> 2 Hombre 18
#> 3 Mujer 20
#> 4 Hombre 18
#> 5 Hombre 18
#> 6 Hombre 18
#> 7 Mujer 19
#> 8 Hombre 18
#> 9 Hombre 18Esta construcción se puede usar también para reordenar las columnas de un data frame; por ejemplo:
d.f1=d.f1[c("Edad","Hermanos","Sexo")]
d.f1
#> Edad Hermanos Sexo
#> 1 17 2 Hombre
#> 2 18 0 Hombre
#> 3 20 0 Mujer
#> 4 18 1 Hombre
#> 5 18 1 Hombre
#> 6 18 1 Hombre
#> 7 19 0 Mujer
#> 8 18 1 Hombre
#> 9 18 2 HombreEl paquete dplyr incluye la función select que amplía las posibilidades para especificar las variables que queremos extraer de un data frame; por ejemplo:
select(data frame, starts_with("x"))extrae del data frame las variables cuyo nombre empieza con la palabrax.select(data frame, ends_with("x"))extrae del data frame las variables cuyo nombre termina con la palabrax.select(data frame, contains("x"))extrae del data frame las variables cuyo nombre contiene en algún sitio la palabrax.
Veamos algunos ejemplos:
library(dplyr)
iris_Petal=select(iris, starts_with("Petal"))
head(iris_Petal, 4)
#> Petal.Length Petal.Width
#> 1 1.4 0.2
#> 2 1.4 0.2
#> 3 1.3 0.2
#> 4 1.5 0.2
iris_Length=select(iris, ends_with("Length"))
head(iris_Length, 4)
#> Sepal.Length Petal.Length
#> 1 5.1 1.4
#> 2 4.9 1.4
#> 3 4.7 1.3
#> 4 4.6 1.5Para más información sobre otras habilidades de la función select, podéis consultar su Ayuda.
Como podéis constatar, hay muchas maneras de extraer una misma subtabla de un data frame dado; veamos una última posibilidad, usando la función subset. La instrucción
subset(data frame, condición, select=columnas)extrae del data frame las filas que cumplen la condición y las columnas especificadas en el select; si queremos todas las filas, no hay que especificar ninguna condición, y si queremos todas las columnas, no hace falta especificar el parámetro select.
Así, si del data frame iris queremos extraer un data frame formado sólo por las variables numéricas de las plantas de especie virginica, podemos usar la instrucción siguiente:
iris.vir=subset(iris, Species=="virginica", select=1:4)
head(iris.vir, 5)
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 101 6.3 3.3 6.0 2.5
#> 102 5.8 2.7 5.1 1.9
#> 103 7.1 3.0 5.9 2.1
#> 104 6.3 2.9 5.6 1.8
#> 105 6.5 3.0 5.8 2.2Fijaos en que las variables en la condición que usamos para seleccionar las filas se especifican con su nombre, sin añadir antes el nombre del data frame
Las filas de este data frame han heredado los identificadores del data frame iris. Si esto nos molesta, los podemos cambiar.
rownames(iris.vir)=1:length(rownames(iris.vir))
head(iris.vir, 5)
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 1 6.3 3.3 6.0 2.5
#> 2 5.8 2.7 5.1 1.9
#> 3 7.1 3.0 5.9 2.1
#> 4 6.3 2.9 5.6 1.8
#> 5 6.5 3.0 5.8 2.26.7 Cómo aplicar una función a las variables de un data frame
La mejor manera de aplicar una función a todas las columnas de un data frame en un solo paso es por medio de la instrucción
sapply(data frame, FUN=función)A modo de ejemplo, vamos a aplicar algunas funciones a las columnas numéricas del data frame iris:
- Para calcular las medias de sus variables numéricas:
sapply(iris[1:4], FUN=mean)
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 5.843333 3.057333 3.758000 1.199333- Para calcular las sumas de sus variables numéricas
sapply(iris[1:4], FUN=sum)
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 876.5 458.6 563.7 179.9- Para calcular las normas euclídeas de sus variables numéricas
f=function(x){sqrt(sum(x^2))}
sapply(iris[1:4], FUN=f)
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 72.27621 37.82063 50.82037 17.38764Es conveniente añadir en el argumento de sapply el parámetro na.rm=TRUE, sin el cual el valor que devolverá la función para las columnas que contengan algún NA será NA. Veamos un ejemplo:
D=data.frame(V1=c(1,2,NA,3), V2=c(2,5,2,NA))
D
#> V1 V2
#> 1 1 2
#> 2 2 5
#> 3 NA 2
#> 4 3 NA
sapply(D, FUN=mean)
#> V1 V2
#> NA NA
sapply(D, FUN=mean, na.rm=TRUE)
#> V1 V2
#> 2 3A menudo querremos aplicar una función a variables de un data frame clasificadas por los niveles de un, o más de un, factor; esto se puede hacer con la instrucción aggregate, cuya sintaxis básica es
aggregate(variable(s)~factor(es), data=data frame, FUN=función)El resultado será un data frame
Por ejemplo, si queremos calcular las medias de las longitudes de los pétalos de las flores de cada una de las tres especies representadas en la tabla iris, podemos entrar la instrucción siguiente:
aggregate(Petal.Length~Species, data=iris, FUN=mean, na.rm=TRUE)
#> Species Petal.Length
#> 1 setosa 1.462
#> 2 versicolor 4.260
#> 3 virginica 5.552Hemos añadido na.rm=TRUE dentro del aggregate para que no tenga en cuenta los NA al calcular la media, por si acaso. Observad que el resultado es un data frame con variables Species y Petal.Length. Cada fila corresponde a un nivel del factor Species.
Si queremos aplicar la función a más de una variable, tenemos que agruparlas a la izquierda de la tilde con cbind.
aggregate(cbind(Petal.Length, Petal.Width)~Species, data=iris, FUN=mean)
#> Species Petal.Length Petal.Width
#> 1 setosa 1.462 0.246
#> 2 versicolor 4.260 1.326
#> 3 virginica 5.552 2.026Si queremos separar las variables mediante más de un factor, tenemos que agruparlos a la derecha de la tilde con signos de suma, +. Veamos un ejemplo. El paquete alr4 contiene la tabla de datos Rateprof, con los resultados globales de la evaluación de un grupo de profesores universitarios por parte de sus estudiantes. Algunas de sus variables son: gender, el sexo del profesor; pepper, que indica si en las encuestas se le ha considerado mayoritariamente atractivo o no; y clarity y easiness, que valoran, entre 1 y 5, la claridad de exposición y la accesibilidad del profesor, respectivamente. Vamos a calcular las medias de estas dos últimas variables agrupándolas por sexo y atractivo.
library(alr4)
aggregate(cbind(clarity,easiness)~gender+pepper, data=Rateprof, FUN=mean)
#> gender pepper clarity easiness
#> 1 female no 3.341391 3.147606
#> 2 male no 3.451456 2.999056
#> 3 female yes 4.345082 3.599128
#> 4 male yes 4.371824 3.689741Observamos que tanto la claridad como la accesibilidad medias de los profesores atractivos de ambos sexos son considerablemente mayores que las de sus colegas considerados no atractivos. Además, en promedio, se considera a los profesores no atractivos menos accesibles que a las profesoras no atractivas, y a los profesores atractivos más accesibles que a las profesoras atractivas.
6.8 Cómo añadir las variables de un data frame al entorno global
Hasta ahora, cada vez que queríamos referirnos a una variable de un data frame, teníamos que escribir el nombre del data frame seguido de $ y el nombre de la variable. Aplicando attach a un data frame, hacemos que R entienda sus variables como globales y que las podamos usar por su nombre, sin necesidad de añadir delante el nombre del data frame y el signo $; esto puede ser útil para no tener que escribir mucho.
A modo de ejemplo, vamos a añadir las variables del data frame iris al entorno global de R. Comprobemos en primer lugar que no tenemos definida ninguna variable llamada Petal.Length:
Petal.Length
#> Error in eval(expr, envir, enclos): objeto 'Petal.Length' no encontradoAhora sí que la habrá:
attach(iris)
Petal.Length[1:30]
#> [1] 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 1.5 1.6 1.4 1.1 1.2 1.5 1.3 1.4 1.7
#> [20] 1.5 1.7 1.5 1.0 1.7 1.9 1.6 1.6 1.5 1.4 1.6Si ya hubiera existido una variable definida con el mismo nombre que una variable del data frame al que aplicamos attach, hubiéramos obtenido un mensaje de error al ejecutar esta función, y no se hubiera reescrito la variable global original.
La función detach devuelve la situación original, eliminando del entorno global las variables del data frame.
detach(iris)
Petal.Length
#> Error in eval(expr, envir, enclos): objeto 'Petal.Length' no encontrado6.9 Guía rápida de funciones
data()produce una lista con los objetos de datos a los que tenemos acceso.Viewmuestra el data frame al que se aplica en la ventana de ficheros.headaplicado a un data frame y un número n, nos muestra las n primeras filas del data frame (por defecto, 6).tailaplicado a un data frame y un número n, nos muestra las n últimas filas del data frame (por defecto, 6).strda la estructura global de un objeto de datos.namessirve para obtener un vector con los nombres de las columnas de un data frame, y también para modificar estos nombres.rownamessirve para obtener un vector con los identificadores de las filas de un data frame, y también para modificar estos identificadores.dimnamessirve para obtener unalistformada por el vector de los identificadores de las filas y el vector de los nombres de las columnas de un data frame, y también para modificar estos vectores simultáneamente.dimda el número de filas y el número de columnas de un data frame.read.tablepermite importar un fichero externo en formato texto simple en un data frame. Algunos parámetros importantes:sep: sirve para indicar los separadores de las columnas.header: sirve para indicar si el fichero a importar tiene una primera fila con los nombres de las variables o no.dec: sirve para especificar el separador decimal.stringsAsFactors: igualado aFALSE, impone que las columnas de palabras se importen como tales, y no como factores.encoding: indica la codificación de alfabeto del fichero externo.
write.table(data frame, file="fichero")exporta el data frame al fichero externo.data.framecrea un data frame con los vectores a los que se aplica. Algunos parámetros importantes:rownames: sirve para especificar los identificadores de las filas.stringsAsFactors: como enread.table.
fixabre un objeto de datos en el editor de datos.paste(x,y,..., sep="separador")pega los vectoresx,y… entrada a entrada, usando como separador el valor del parámetrosep.as.charactertransforma todos los datos de un objeto en palabras.as.integertransforma todos los datos de un objeto en números enteros.as.numerictransforma todos los datos de un objeto en números reales.as.romantransforma números enteros en números romanos.droplevelsborra todos los niveles sobrantes de todos los factores de un data frame.select, del paquetedplyr, permite definir un data frame con todas las variables de un data frame que empiecen por una secuencia de letras dada (con el parámetrostarts_with), que terminen por una secuencia de letras dada (con el parámetroends_with), o que contengan una secuencia de letras dada (con el parámetrocontains).subset(data frame, condición, select=columnas)define un data frame con las filas del data frame que cumplen lacondicióny las columnas especificadas en el parámetroselect.sapply(data frame, FUN=función)aplica lafuncióna las columnas de un data frame.aggregatesirve para aplicar una función a una o varias variables de un data frame agrupando sus entradas por los niveles de uno o varios factores.attachañade las variables de un data frame al entorno global de R.detachdeshace el efecto deattach.clipboard(), del paquete readr , da el contenido del Portapapeles.
6.10 Ejercicios
Test
(1) El data frame CO2 es uno de los que lleva predefinidos R. ¿Qué vale la variable conc para la observación correspondiente a la cuadragésima quinta fila? Dad su valor, no cómo lo habéis obtenido.
(2) Suponed que tenéis un data frame llamado Datos1. Dad una instrucción que muestre su estructura.
(3) Suponed que tenéis un data frame llamado Datos1 que tiene una variable llamada Temp de tipo entero (int). Dad una instrucción, lo más sencilla posible, que redefina este data frame de forma que ahora su variable “Temp” sea de tipo numérico (num).
(4) El data frame DNase es uno de los que lleva predefinidos R. Cread un nuevo data frame añadiéndole una nueva fila donde la variable Run tome el valor 5, la variable conc tome el valor 0.5 y la variable density tome el valor 2.3. ¿Qué vale la media, redondeada a 4 cifras decimales, de los valores de la variable density en las filas del data frame resultante cuyo valor de la variable Run sea 5? Tenéis que dar el resultado final, no cómo lo habéis obtenido.
(5) Definid un data frame con la tabla contenida en el url http://aprender.uib.es/Rdir/heartatk4R.txt y calculad el valor máximo de la variable AGE entre los individuos cuya variable SEX valga F. Tenéis que dar el resultado final, no cómo lo habéis obtenido.
(6) Definid un data frame con la tabla contenida en el url http://aprender.uib.es/Rdir/ESD.txt y calculad, redondeado a 2 cifras decimales, el valor medio de la variable MB. Tenéis que dar el resultado final, no cómo lo habéis obtenido.
(7) Definid un data frame con la tabla contenida en el url http://aprender.uib.es/Rdir/ESD.txt y dad el valor de la variable Year para el cual el valor medio de la variable BH sea mayor. Tenéis que dar el resultado final, no como lo habéis obtenido.
(8) Dad una instrucción que use la función read.table para importar en un data frame llamado Datos un fichero llamado “datos.txt” situado en la carpeta de trabajo de R. Las separaciones entre columnas en este fichero son tabuladores y tiene una primera fila con los nombres de las columnas.
Ejercicio
La tabla de datos pulse.txt que encontraréis en http://aprender.uib.es/Rdir/pulse.txt recoge una serie de informaciones de tipo general (altura, peso, sexo, si corren, si fuman y su nivel de actividad física: 1 si es bajo, 2 si es moderado y 3 si es alto) sobre algunos estudiantes matriculados en un curso de estadística de la Universidad Estatal de Pensilvania hace unos años. A estos estudiantes se les pidió que lanzasen una moneda al aire: a los que sacaron cara, se les hizo correr un minuto sin moverse del sitio, y los que sacaron cruz, descansaron un minuto. Todos los estudiantes (tanto los que corrieron como los que no) midieron sus pulsaciones por minuto antes y después de este minuto de ejercicio o descanso, y estas medidas también aparecen en esta tabla (en las variables PuBefor y PuAfter, respectivamente).
¿Cuántos estudiantes tomaron parte en este estudio? ¿Cuántos son hombres y cuántas mujeres?
Calculad el porcentaje medio de variación en el número de pulsaciones tras el minuto de ejercicio o descanso de los estudiantes que corrieron (se indica con el valor
yesen la variableRan.) y de los que no. ¿Hay mucha diferencia?Calculad el porcentaje medio de incremento en el número de pulsaciones tras el minuto de ejercicio sólo para los estudiantes que corrieron, pero ahora distinguiendo los hombres de las mujeres. ¿Cuál de los dos incrementos medios es mayor?
Calculad el porcentaje medio de incremento en el número de pulsaciones tras el minuto de ejercicio para los estudiantes que corrieron, pero ahora distinguiendo los estudiantes que fuman de los que no. ¿Cuál de los dos incrementos medios es mayor?
Calculad el número medio de pulsaciones antes del minuto de ejercicio o descanso de todos los estudiantes, separados según su nivel de actividad física. ¿Se observa alguna diferencia significativa?
Respuestas al test
(1) 250
(2) str(Datos1)
(3) Datos1$Temp=as.numeric(Datos1$Temp)
(4) 0.7985
(5) 102
(6) 133.97
(7) -1850
(8) Datos=read.table("datos.txt",sep="\t",header=TRUE)